Obecnie metody wykorzystujące sztuczną inteligencję (ang. Artificial Intelligence, AI) rewolucjonizują rynek diagnostyki medycznej na całym świecie.
AI jest wykorzystywana do analizy wyników w różnych dziedzinach medycyny, takich jak patomorfologia, kardiologia i onkologia. Dotychczas nie wprowadzono żadnego rozwiązania opartego na AI do diagnostyki mikrobiologicznej. Obecnie stosowane metody diagnostyki mikrobiologicznej obejmują standardowe metody hodowlane (fenotypowe), testy serologiczne oraz molekularne. Metody te mają jednak swoje wady, takie jak długi czas oczekiwania na wyniki lub ograniczoną dostępność.
Nowe, innowacyjne rozwiązanie z Uniwersytetu Jagiellońskiego może wspomóc diagnostykę mikrobiologiczną z wykorzystaniem algorytmów AI. Zostało opracowane przez zespół naukowy pod kierunkiem prof. dr hab. Moniki Brzychczy‑Włoch, kierownika Zakładu Molekularnej Mikrobiologii Medycznej UJ CM, oraz dr. hab. Bartosza Zielińskiego, prof. UJ z Wydziału Matematyki i Informatyki UJ. Od dłuższego czasu pracują oni nad zastosowaniem metod głębokiego uczenia maszynowego (ang. deep learning) do klasyfikacji i identyfikacji pojedynczych gatunków bakterii z mieszanin wielogatunkowych, obrazowanych na zdjęciach mikroskopowych. Nowatorskie rozwiązanie jest chronione międzynarodowym zgłoszeniem patentowym pt. „A method and a system for identifying polyculture bacteria on microscopic images using deep learning”, nr EP 22461550.0. Umożliwia ono szybką identyfikację mikroorganizmów na zdjęciach z mikroskopów świetlnych.
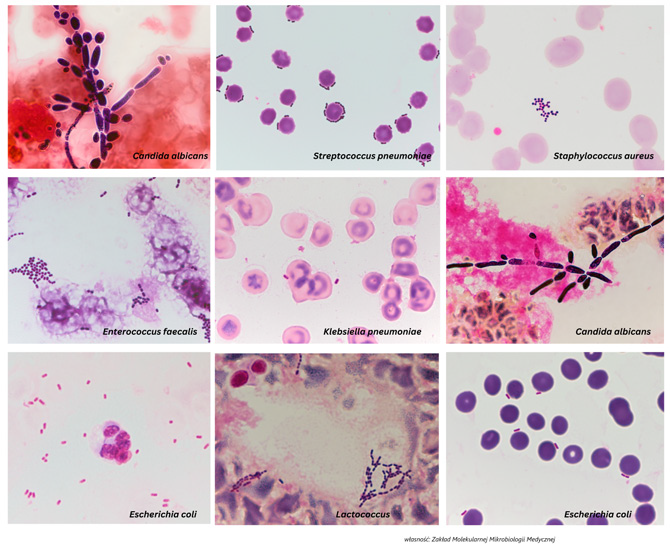
Sepsa- preparaty mikroskopowe z dodatnim psiewem krwi barwiene metodą Grama.
Technologia ta opiera się na głębokich sieciach neuronowych, a jej celem jest wspomaganie pracy mikrobiologów, diagnostów laboratoryjnych i lekarzy. Proces identyfikacji mikroorganizmów przy użyciu klasycznych metod mikrobiologicznych składa się z wielu etapów i może trwać od kilku dni do kilku tygodni. Dzięki zastosowaniu metod uczenia maszynowego wykorzystujących multi‑MIL (ang. deep multiple instances learning) możliwe jest znaczące skrócenie czasu identyfikacji bakterii do poziomu gatunku, co przyspieszy diagnostykę mikrobiologiczną u pacjentów. Powstałe rozwiązanie cechuje się wysoką precyzją (specyficzność identyfikacji taksonomicznej bakterii powyżej 90%). Główne zalety zastosowania tej metody w diagnostyce mikrobiologicznej to m.in. skrócenie czasu identyfikacji drobnoustrojów (o kilka dni), ograniczenie kosztów oraz nakładu pracy diagnostów laboratoryjnych i specjalistów mikrobiologii medycznej, brak konieczności stosowania specjalistycznej aparatury diagnostycznej oraz przyspieszenie wdrożenia skutecznej terapii przeciwdrobnoustrojowej.
W problematykę szybkiej diagnostyki mikrobiologicznej wpisuje się również nowy projekt kierowany przez prof. dr hab. Monikę Brzychczy‑Włoch pt. „Inteligentna walka z sepsą – innowacyjne wykorzystanie sztucznej inteligencji w szybkiej diagnostyce mikrobiologicznej sepsy” (nr FENG.02.07- -IP.05–0068/23) w ramach działania Proof of Concept (PoC) finansowanego z programu Fundusze
Europejskie dla Nowoczesnej Gospodarki (FENG). Dofinansowanie projektu z EU wynosi 643 440,00 zł. Celem projektu jest opracowanie metody, wykorzystującej algorytmy AI do identyfikacji drobnoustrojów na podstawie obrazów z preparatów mikroskopowych, barwionych metodą Grama, wykonanych z rozmazów dodatnich hodowli krwi pacjentów z sepsą. Innowacyjne rozwiązanie opracowane w ramach projektu PoC pozwoli na redukcję kosztów i ograniczenie zużycia zasobów poprzez wyeliminowanie konieczności wykonywania wielu hodowli i testów do identyfikacji patogenów. Co najważniejsze, umożliwi znaczące skrócenie czasu diagnostyki i wdrożenie skutecznej terapii przeciwdrobnoustrojowej. Efektem końcowym projektu będzie opracowanie prototypu programowania komputerowego – MicroBloodID, które na podstawie obrazu z preparatów mikroskopowych, barwionych metodą Grama, dzięki metodom głębokiego uczenia maszynowego, identyfikuje czynnik etiologiczny zakażenia i wygeneruje spersonalizowane zalecenia empirycznej terapii przeciwdrobnoustrojowej.
Ochroną prawną wyżej opisanych rozwiązań Uniwersytetu Jagiellońskiego oraz kompleksową współpracą nauki z biznesem zajmuje się Centrum Transferu Technologii CITTRU. Obecnie Uniwersytet Jagielloński wraz z zespołem naukowym pod kierunkiem Pani prof. dr hab. Moniki Brzychczy‑Włoch we współpracy z zespołem dr hab. Bartosza Zielińskiego, prof. UJ, poszukuje podmiotów zainteresowanych komercyjnym wykorzystaniem opisanych rozwiązań i współpracą w tym zakresie.




Katarzyna Małek‑Ziętek,
dr Klaudia Polakowska
Centrum Transferu Technologii CITTRU


